„Burden of Disease“ durch multiresistente Erreger: Keine Entspannung in Sicht!
DOI: https://doi.org/10.47184/td.2024.04.05Der Anteil von Nachweisen und Infektionen durch multiresistente Erreger, insbesondere durch gramnegative Stäbchenbakterien und Enterokokken, nimmt bundes- und weltweit seit Jahren stetig zu. Die vorliegende Übersicht soll einen Einblick in die aktuelle Situation geben.
Schlüsselwörter: Carbapenem-Resistenz, Carbapenemasen, ESBL, Vancomycin-Resistenz, ESKAPE
Carbapenem-resistente Enterobacterales und Non-Fermenter
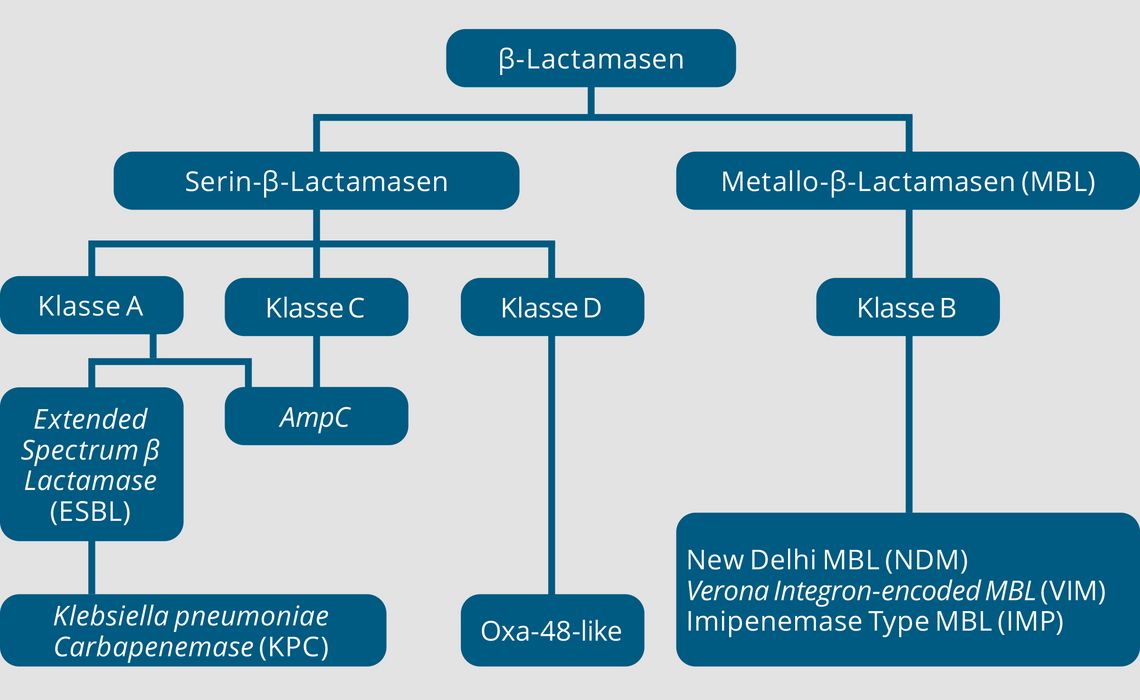
Carbapeneme gelten immer noch als Antibiotika-Klasse mit dem breitesten Wirkspektrum aus der Gruppe der β-Lactame [1]. Ein wichtiger und verbreiteter Mechanismus der Carbapenem-Resistenz bei gramnegativen Bakterien ist die Produktion von erworbenen Carbapenemasen, die gemäß der Ambler-Klassifikation in die Gruppen A, B, C und D eingeteilt werden (Abb. 1; Anmerkung: Gruppe C [AmpC-kodierte Resistenzen] ist meist gegenüber Carbapenemen empfindlich).
Zusätzlich zur Hydrolyse von Carbapenemen sind diese Enzyme mit wenigen Ausnahmen auch gegen die meisten anderen Mitglieder der β-Lactam-Gruppe aktiv. Hauptgrund für das Auftreten von Carbapenemasen war der weitverbreitete Einsatz von Carbapenemen bei der Behandlung schwerer Infektionen, die durch Extended Spectrum Beta Lactamase(ESBL)-produzierende Erreger verursacht werden [2]. Die erste Carbapenemase (IMP) wurde bereits im Jahr 1990 in Japan beschrieben. Die meisten Carbapenemasen werden durch transposable Plasmide kodiert (u. a. VIM, IMP und NDM), und einige wenige liegen chromosomal vor (beispielsweise L1 bei Stenotrophomonas maltophilia). Bei der zunehmenden Fokussierung auf Carbapenemase-bildende Infektionserreger darf jedoch nicht vergessen werden, dass bei der Mehrzahl dieser Erreger keine der typischen Carbapenemase-kodierenden Gene gefunden wurden, das Enzym aber dennoch nachgewiesen werden kann – folglich muss man hier seltene Gene vermuten [3].
Risikokonstellationen für die Infektion durch Carbapenem-resistente Erreger sind vorangegangene Antibiotika-Therapien, ein Aufenthalt auf einer Intensivstation, ggf. mit künstlicher Beatmung, Komorbiditäten mit chronischen Erkrankungen, Reisen in „Carbapenemase“-Endemiegebiete und eine bestehende Besiedelung mit Carbapenemase-bildenden Erregern [4]. Aufgrund der deutlich eingeschränkten Therapiemöglichkeiten bei Vorliegen eines Erregers mit Carbapenemase-Aktivität ist eine Infektion mit deutlich erhöhter Letalität und verlängerter Hospitalisierung verbunden: In einer Fallkontrollstudie über nosokomiale Infektionen durch NDM-1-positive Enterobacterales betrug die Krankenhausmortalität der Betroffenen 55,3 versus 17,0 % bei Infektionen durch empfindliche Stämme; eine umfängliche Übersicht zu Patient:innen mit Pneumonien durch Stenothrophomonas maltophilia mit intrinsischer Carbapenemase-Aktivität zeigte eine Mortalität je nach Grunderkrankung zwischen 22 und 77 % (die höchste Mortalität wurde bei Tumorpatient:innen mit Blutstrominfektionen gefunden) [5, 6].
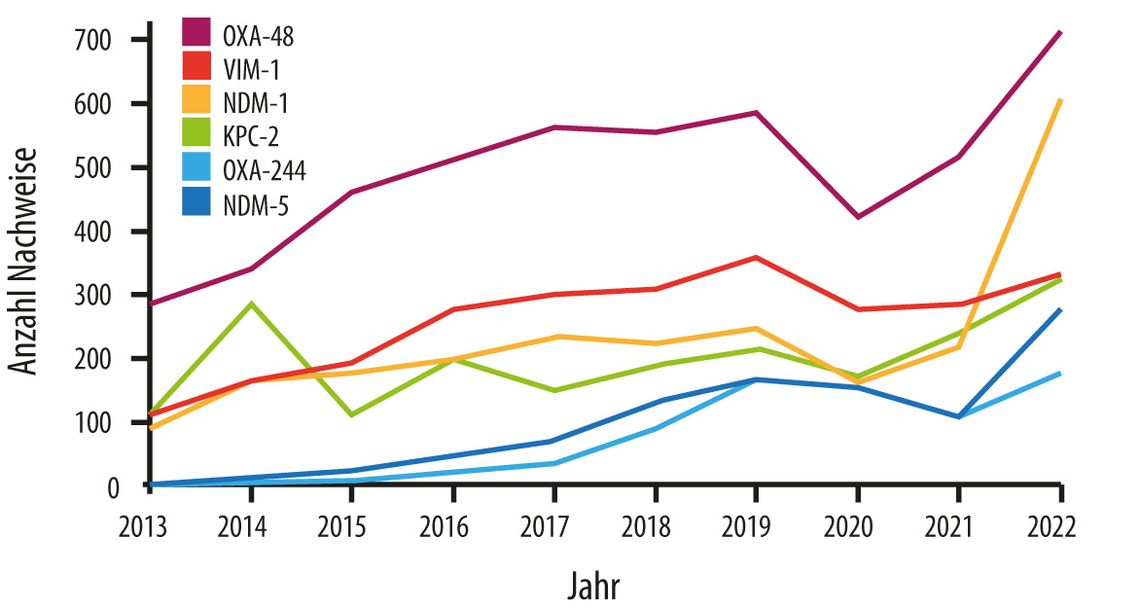
Die Einsendungen an das Nationale Referenzzentrum zum Nachweis oder zur Bestätigung einer Carbapenemase haben sich von 2012 bis 2022 nahezu verdreifacht (2012: n = 3.883; 2022: n = 9.548), was tendenziell der epidemischen Resistenzverbreitung von Carbapenemasen weltweit entspricht [7, 8]. Am häufigsten erfolgte der Nachweis von Oxa-48 bei Klebsiella (Kl.) pneumoniae, gefolgt von den Metallo-β-Lactamasen NDM-1 – ebenfalls am häufigsten bei Kl. pneumoniae – und VIM-1 bei Enterobacter cloacae (Abb. 2).
Eine Herausforderung beim Nachweis einer Infektion durch Carbapenemase-Bildner stellt zweifelsohne die Therapie dar: Selbst neuere β-Lactam/β-Lactamase-Inhibitor-Kombinationen (Ceftazidim-Avibactam, Imipenem-Relebactam, Meropenem-Vaborbactam) sind zur Behandlung nur eingeschränkt geeignet. Tab. 1 bietet eine Übersicht über die aktuellen Therapieempfehlungen gemäß der Infectious Disease Society of America (IDSA) bzw. der European Society of Clinical Microbiology and Infectious Diseases (ESCMID) [9, 10].
Tab. 1: Erstlinientherapie gemäß ESCMID-/ IDSA-Leitlinie [9, 10] bei Vorliegen von Carbapenem-resistenten Stämmen.
Für die Mehrzahl der Empfehlungen existieren Daten aus kleinen, retrospektiven Studien (geringe Evidenz).
Erreger | Indikationen/Anmerkungen | Therapie |
|---|---|---|
Carbapenem-resistenter Pseudomonas aeruginosa (Carbapenem-Resistenz nicht spezifiziert bzw. anderer Mechanismus als Carbapenemase-Bildung)
Carbapenem-resistente P. aeruginosa mit Nachweis einer Metallo-β-Lactamase | Schwere Infektionen
Unkomplizierte Harnwegsinfektionen
Schwere Infektionen |
Aminoglykoside (Einmaldosis Tobramycin, Amikacin)
Cefiderocol |
Carbapenem-resistenter Acinetobacter baumannii (CRAB) | CRAB: generell Kombinationstherapie zweier wirksamer Antibiotika empfohlen
Ambulant erworbene Pneumonie/Beatmungsassozierte Pneumonie |
|
Carbapenem-resistente Enterobacterales ohne Nachweis einer Carbapenemase
Carbapenem-resistente Enterobacterales mit Nachweis von KPC, Oxa-48
Carbapenem-resistente Enterobacterales mit Nachweis einer Metallo-β-Lactamase (einschl. NDM) | Infektionen (außer Harnwegsinfektionen)
Schwere Infektionen
Schwere Infektionen
Haut-Weichteilinfektionen mit Carbapenem-resistenten Enterobacterales
Unkomplizierte Harnwegsinfektionen bei Carbapenem-resistenten Enterobacterales |
Hinweis: Imipenem/Cilastin-Relebactam bei Nachweis von Oxa-48 unwirksam
Tigecyclin
Colistin |
Carbapenem-resistenter Stenotrophomonas maltophilia | EUCAST-Breakpoint vorhanden Nach Guidance Document (EUCAST)
Keine EUCAST-Breakpoints vorhanden
Schwere Infektionen in Kombinationstherapie |
|
Vancomycin-resistente Enterokokken
Glykopeptid-(Vancomycin-)resistente Enterokokken (VRE) – insbesondere Enterococcus (E.) faecium – werden zur Gruppe der ESKAPE-Bakterien gezählt, die sich aufgrund von Resistenzen durch eine deutlich eingeschränkte Therapierbarkeit mit erhöhter Morbidität und Mortalität auszeichnen [11] (ESKAPE: E = Enterococcus faecium / S = Staphylococcus aureus / K = Klebsiella pneumoniae / A = Acinetobacter baumannii / P = Pseudomonas aeruginosa / E = Enterobacter spp.).
Der Resistenzmechanismus der Vancomycin-Resistenz beruht auf den erworbenen (meist Plasmid-kodierten) Resistenzgenen vanA / vanB / vanD / vanE / vanG / vanL / vanM und vanN, wobei vanA und vanB weltweit am häufigsten vorkommen [12]. Das vanA-Gen kodiert für eine kombinierte Resistenz gegenüber Vancomycin und Teicoplanin (High-Level-Resistenz Vancomycin MIC > 32 mg/l / Teicoplanin MIC > 16 mg/l), während der vanB-Phänotyp durch eine variable Vancomycin-Resistenz charakterisiert ist – mit Empfindlichkeit gegenüber Teicoplanin. VRE zeigen darüber hinaus intrinsische Resistenzen gegenüber Cephalosporinen (PBP5-Mutation), Lincosamiden, Trimethoprim-Sulfamethoxazol und erworbenen Resistenzen gegenüber Aminoglykosiden (High-Level-Resistenz) und Ampicillin.
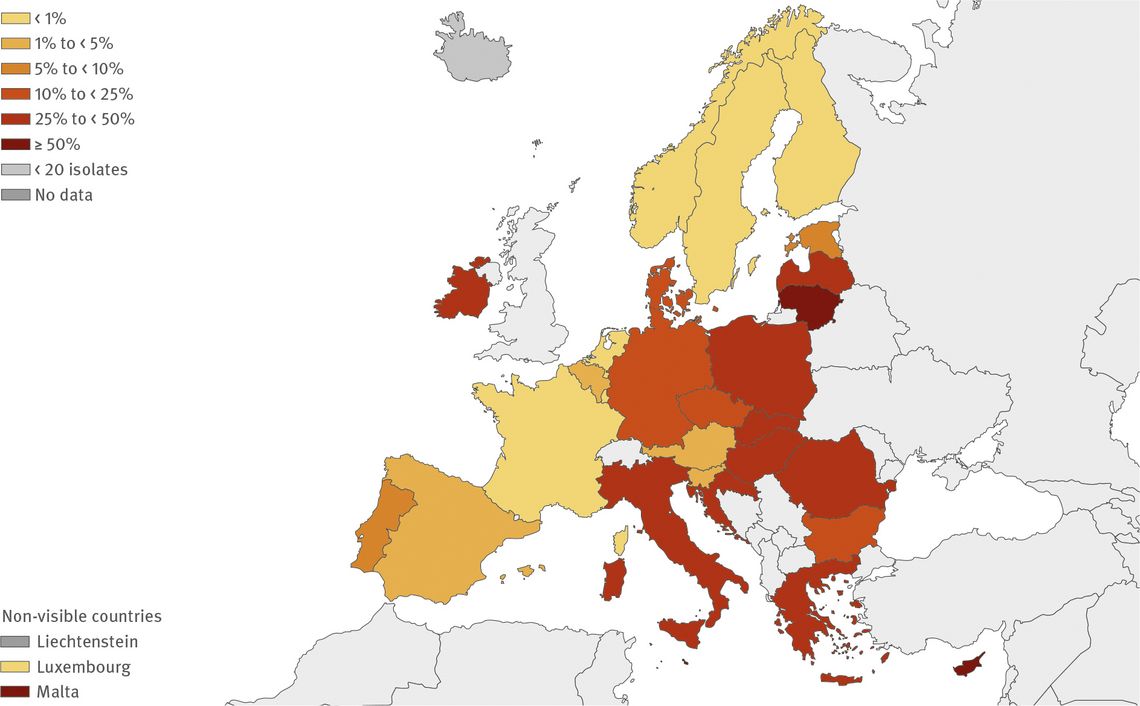
Im Hinblick auf die Epidemiologie von Vancomycin-resistenten (Vr.) E. faecium gehört die BRD laut Surveillance-Report des European Center for Disease Control (ECDC) zu den Hochendemiegebieten: Der Anteil an VRE bei invasiven Infektionen beträgt mittlerweile um die 25 % und wird damit nur noch von den süd- und südosteuropäischen Staaten übertroffen (Abb. 3; [13]); innerhalb der BRD existiert ein Nord-Süd-Gefälle mit Vr. E. faecium in Bayern von bis zu 40 % (2019; [13]).
Die hohe Prävalenz von Vr. E. faecium in der BRD ist nicht wirklich über einen hohen Verbrauch von Antibiotika erklärbar, da die BRD zu den Niedrig-Antibiotika-Verbrauchenden in der EU zählt [14] (Hypothese: „Resistenzentstehung ist das Ergebnis der Gesamtheit der Antibiotika-Verbräuche!“).
Die Morbidität und Mortalität hat sich durch schwere VRE-Infektionen weltweit von 2007 bis 2015 verdoppelt, was die ECDC dazu veranlasst, zu einer verbesserten Surveillance sowie besseren Untersuchungen der klonalen Diversitäten und Risikofaktoren im Zusammenhang mit VRE aufzurufen.
Wie bereits oben angedeutet, zeigen bei den Vancomycin-Resistenzmechanismen die E. faecium-Stämme mit vanA- oder vanB-kodierten Resistenzen die höchste Prävalenz. Hierbei hat sich in der BRD und in benachbarten europäischen Ländern in den vergangenen Jahren ein Shift von vanA zu vanB bei Vr. E. faecium vollzogen, der mit bestimmten genetischen Clustern einhergeht [12, 16]: So dominieren in der BRD die Stammtypen (ST) ST117 (2022: 53 % aller Isolate am Nationalen Referenzzentrum) und ST80 (38 %), gefolgt von ST1299 (4 %) und ST78 (3 %). Die Gründe für die Ausbreitung bestimmter Stammtypen sind nicht bekannt.
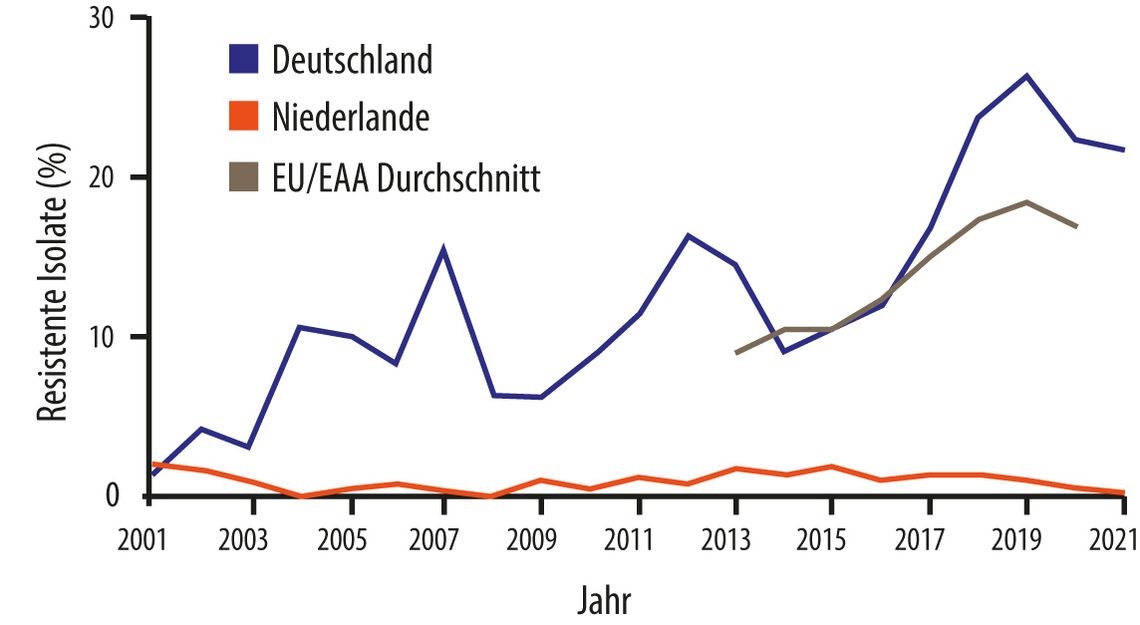
Aufgrund der Zunahme von schweren Infektionen durch VRE (Blutstrominfektionen, insbesondere im Zusammenhang mit Devices [17–19]) wurden auch in der BRD nationale VRE-Infektionspräventionsmaßnahmen empfohlen [20]. Die deutschen Richtlinien orientieren sich mit ihren Empfehlungen an der epidemiologischen Situation des jeweiligen Krankenhauses bzw. der Region und an Risikogruppen; im Gegensatz dazu verfolgen die holländischen Richtlinien eine Search and Detect-Strategie [21], die bekanntermaßen schon bei Methicillin-resistenten S. aureus-Stämmen erfolgreich war (Abb. 4; [22]).