CHIP: Molekulare Mechanismen, Diagnostik und therapeutische Implikationen: Schlüsselphänomen bei Alterung und kardiovaskulären Erkrankungen
DOI: https://doi.org/10.47184/td.2025.01.03Somatische Mutationen in hämatopoetischen Stamm- oder Vorläuferzellen, die zu einer Aktivierung und Proliferation dieser Zellen führen, ohne dass bei den Betroffenen eine hämatologische Erkrankung in Erscheinung tritt, werden als klonale Hämatopoese unbestimmten Potenzials – kurz CHIP – bezeichnet. Personen, bei denen die Mutationen nachgewiesen werden, haben unter anderem ein erhöhtes Risiko für kardiovaskuläre Erkrankungen.
Schlüsselwörter: Koronare Herzerkrankung, NGS, IL-1β-Inhibitoren, DNMT3A, TET2, ASXL1
Die klonale Hämatopoese unbestimmten Potenzials (Clonal Hematopoiesis of Indeterminate Potential; CHIP) hat sich als zunehmend relevanter Forschungsgegenstand etabliert, der ein besseres Verständnis der Zusammenhänge zwischen Alterung, somatischen Mutationen und der Entwicklung chronischer Erkrankungen ermöglicht. CHIP beschreibt das Auftreten somatischer Mutationen in hämatopoetischen Stamm- oder Vorläuferzellen, die zu einer klonalen Expansion dieser Zellen führen, ohne dass eine hämatologische Erkrankung im klassischen Sinne manifest wird [1, 2]. Das Auftreten von CHIP-Mutationen steigt mit dem Lebensalter an und zeigt eine Prävalenz von 30 bis 40 % bei über 70-jährigen Individuen, während die Mutationen in der Gesamtbevölkerung mit etwa 10 bis 20 % deutlich seltener vorkommen. Bemerkenswert ist, dass CHIP nicht nur mit hämatologischen Neoplasien assoziiert ist, sondern sich als unabhängiger Risikofaktor für nichthämatologische Erkrankungen – insbesondere kardiovaskuläre Erkrankungen (CVD) – etabliert hat. Bei Individuen mit koronarer Herzerkrankung trägt jeder Dritte mindestens eine CHIP-Mutation [3, 4]. Die steigende Aufmerksamkeit für CHIP ergibt sich aus seiner Schlüsselrolle bei der Förderung chronischer Entzündungsprozesse, die sowohl die Pathogenese als auch die Progression von CVD beeinflussen.
WHO 2022: Kategorisierung und Bedeutung
Die Weltgesundheitsorganisation (WHO) hat 2022 54 Gene mit CHIP assoziiert (Tab. 1).
Tab. 1: Die Liste von CHIP-definierenden Genen der Weltgesundheitsorganisation (WHO) von 2022 umfasst folgende 54 Mutationen (alphabetisch sortiert). Ergänzt wurden bekannte Effekte und assoziierte Pathways. Die häufigsten zehn CHIP-Mutationen wurden gekennzeichnet [5].
Gen | Effekt | Assoziierte Pathways |
|---|---|---|
ASXL1 | Chromatin-Modifikation | Chromatin-Remodelling |
BCOR | Chromatin-Modifikation | Transkriptionskontrolle |
BCORL1 | Chromatin-Modifikation | Transkriptionskontrolle |
BRAF | Signaltransduktion | MAPK-Signalweg |
BRCC3 | DNA-Reparatur | DNA-Reparatur |
CALR | Signaltransduktion | Signaltransduktion |
CBL | Signaltransduktion | Ubiquitinierung |
CEBPA | Transkriptionsfaktor | Transkriptionsregulation |
CRBBP | Chromatin-Modifikation | Chromatin-Acetylierung |
CSF1R | Signaltransduktion | Koloniestimulierende Faktoren |
CSF3R | Signaltransduktion | Koloniestimulierende Faktoren |
CTCF | Chromatin-Modifikation | Chromatin-Stabilität |
CUX1 | Chromatin-Modifikation | Transkriptionsregulation |
DNMT3A | Epigenetische Regulation | DNA-Methylierung |
ETV6 | Transkriptionsfaktor | Signaltransduktion |
EZH2 | Chromatin-Modifikation | Chromatin-Modifikation |
GATA2 | Chromatin-Modifikation | Hämatopoese |
GNAS | Signaltransduktion | Signaltransduktion |
GNB1 | Signaltransduktion | Signaltransduktion |
IDH1 | Metabolismus | Zellulärer Metabolismus |
IDH2 | Metabolismus | Zellulärer Metabolismus |
JAK2 | Signaltransduktion | JAK/STAT |
JAK3 | Signaltransduktion | Signaltransduktion |
KDM6A | Chromatin-Modifikation | Epigenetische Regulation |
KIT | Signaltransduktion | Signaltransduktion |
KMT2A | Signaltransduktion | Signaltransduktion |
KRAS | Signaltransduktion | RAS/MAPK |
MPL | Signaltransduktion | NF-κB-Signalweg |
MYD88 | Transkriptionsfaktor | Signaltransduktion |
NOTCH1 | Signaltransduktion | Transkriptionskontrolle |
NRAS | Signaltransduktion | RAS/MAPK |
PHF6 | RNA-Splicing | GPI-Anker-Synthese |
PIGA | Tumorsuppressor | RNA-Splicing |
PPM1D | DNA-Reparatur | p53-Achse |
PRPF40B | Chromatin-Modifikation | Tumorsuppressor |
PTEN | Transkriptionsfaktor | Chromatin-Stabilität |
PTPN11 | Signaltransduktion | RAS/MAPK |
RAD21 | Chromatin-Modifikation | Transkriptionskontrolle |
RUNX1 | Chromatin-Modifikation | Chromatin-Stabilität |
SETBP1 | Chromatin-Modifikation | Chromatin-Stabilität |
SF1 | Signaltransduktion | Cohesin-Komplex |
SF3A1 | RNA-Splicing | Signaltransduktion |
SF3B1 | RNA-Splicing | Spliceosom |
SMC1A | Chromatin-Modifikation | Spliceosom |
SMC3 | Signaltransduktion | Chromatin-Stabilität |
SRSF2 | RNA-Splicing | Spliceosom |
STAG2 | RNA-Splicing | Signaltransduktion |
STAT3 | Chromatin-Modifikation | Spliceosom |
TET2 | Epigenetische Regulation | Hydroxymethylierung |
TP53 | Tumorsuppressor | p53-Signalweg |
U2AF1 | RNA-Splicing | Spliceosom |
U2AF2 | Unbekannt | Chromatin-Stabilität |
WT1 | Unbekannt | Unbekannt |
ZRSR2 | Unbekannt | Unbekannt |
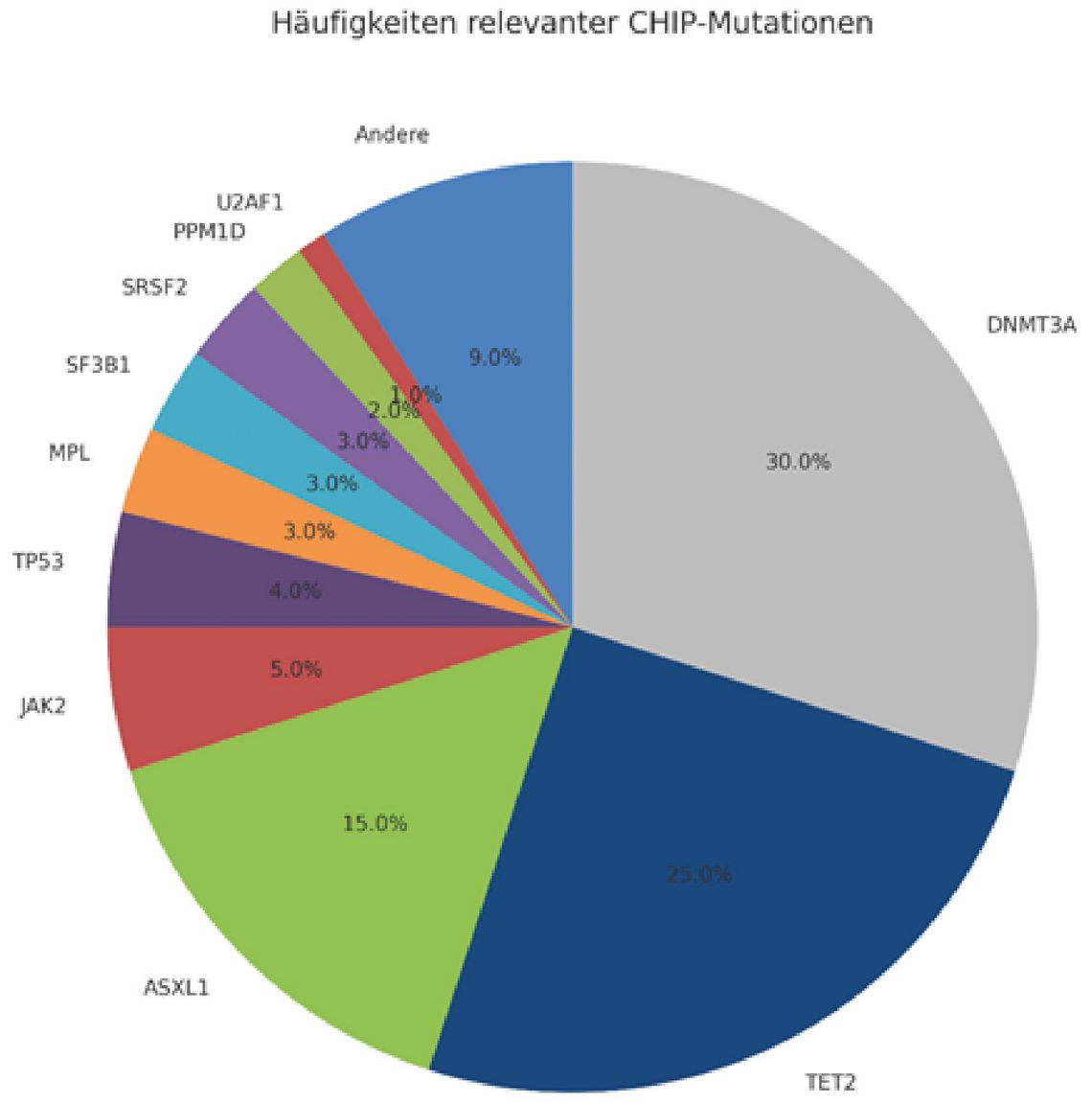
Diese lassen sich in verschiedene funktionelle Gruppen einteilen, die unterschiedliche Mechanismen der Krankheitsentstehung beeinflussen und potenzielle Therapieansätze aufzeigen [6]. Zu den zentralen Akteuren gehören epigenetische Regulatorgene wie DNMT3A, TET2 und ASXL1, die mehr als zwei Drittel aller CHIP-Mutationen ausmachen (Abb. 1).
Signaltransduktionsgene wie JAK2 spielen ebenfalls eine wichtige Rolle und sind mit spezifischen pathologischen Prozessen assoziiert. Eine dritte Gruppe umfasst Gene, die an der DNA-Reparatur und Chromatin-Modifikation beteiligt sind, und eine vierte Gruppe betrifft solche, die die Differenzierung und Funktion von Immunzellen beeinflussen. Trotz dieser umfassenden Klassifikation sind die Mechanismen, die der Wirkung vieler CHIP-assoziierter Gene zugrunde liegen, bisher nur unzureichend verstanden [7].
Pathophysiologie
Die Pathophysiologie von CHIP basiert auf der klonalen Expansion mutierter hämatopoetischer Stamm- oder Vorläuferzellen im Knochenmark, die einen Selektionsvorteil gegenüber ihren nichtmutierten Gegenstücken erlangen [1, 2, 8]. Dieser Selektionsvorteil resultiert häufig aus einer Resistenz gegenüber apoptotischen Signalen und einer verbesserten Fähigkeit zur Proliferation in proinflammatorischen Mikroumgebungen. Inflammation nimmt eine zentrale Rolle in der Krankheitsprogression ein, da die durch CHIP-Mutationen betroffenen Zellen die Produktion proinflammatorischer Mediatoren verstärken, die sowohl systemische als auch lokale Entzündungsreaktionen fördern. Dabei beeinflussen CHIP-mutierte Zellen ihre Umgebung bereits in der Knochenmarknische mit systemischer Relevanz. Unter den bekannten CHIP-assoziierten Mutationen zeigen insbesondere Veränderungen in TET2 eine besonders starke Verbindung zur gesteigerten Inflammation. TET2-defiziente Zellen sekretieren erhöhte Mengen an Interleukin-1β (IL-1β) und Interleukin-6 (IL-6), zwei zentralen proinflammatorischen Zytokinen, die endothelialen Stress verursachen, die Funktionalität von Endothelzellen beeinträchtigen und die Destabilisierung atherosklerotischer Plaques fördern. Dies erklärt, warum CHIP-Träger:innen ein signifikant erhöhtes Risiko für kardiovaskuläre Ereignisse wie Myokardinfarkt und Schlaganfall aufweisen [9].
Tierexperimentelle Modelle
Tierexperimentelle Modelle konnten belegen, dass TET2-defiziente Makrophagen die Atherogenese durch verstärkte Sekretion von Chemokinen beschleunigen, die weitere Immunzellen rekrutieren und eine entzündungsfördernde Feedbackschleife aufrechterhalten [10]. Die Beteiligung von CHIP an der Progression der Atherosklerose geht jedoch über die Effekte einzelner Gene hinaus. Mutationen in DNMT3A und ASXL1 beeinflussen epigenetische Programme, die den Metabolismus und die Differenzierung hämatopoetischer Zellen modulieren. Diese Veränderungen verstärken nicht nur inflammatorische Prozesse, sondern fördern auch immunsystemassoziierte Signalkaskaden, die zur Verschlechterung der vaskulären Integrität beitragen. Zudem zeigen erste Untersuchungen, dass Splicing-Mutationen bei CHIP die Genexpression inflammatorischer Mediatoren beeinflussen können, was die Komplexität der molekularen Mechanismen weiter erhöht [1–3, 11]. Es bleibt jedoch anzumerken, dass für viele CHIP-assoziierte Mutationen die zugrunde liegenden pathophysiologischen Mechanismen noch nicht vollständig verstanden sind.
Präzision durch Next Generation Sequencing
Die Diagnostik von CHIP stützt sich auf die Analyse somatischer Mutationen mittels moderner, tiefer Sequenzierungstechnologien wie Next Generation Sequencing (NGS). Eine Variantenallelfrequenz (VAF) von ≥ 2 % wurde als diagnostische Schwelle definiert. Dieser Cut-off ist historisch bedingt und mit einer vormals eingeschränkten Sequenziertiefe zu erklären. Es konnte bereits gezeigt werden, dass auch eine VAF von < 2 % relevant für das kardiovaskuläre Risiko ist. Während die Häufigkeit bei Personen unter 50 Jahren bei etwa 2 bis 5 % liegt, steigt sie bei über 80-Jährigen auf bis zu 50 % [1–3]. Neben dem Alter tragen externe Faktoren wie Rauchen, Hyperlipidämie, Stress und metabolische Störungen zur Expansion CHIP-assoziierter Klone bei, indem sie die inflammatorische Mikroumgebung verstärken und den Selektionsdruck zugunsten der klonalen Expansion erhöhen [1, 3, 12].
Klinische Relevanz und therapeutische Ansätze
Die klinische Relevanz von CHIP ergibt sich aus seiner starken Assoziation mit kardiovaskulären Ereignissen und seiner Rolle als unabhängiger Risikofaktor. Retrospektive Analysen von großen antiinflammatorischen Interventionsstudien wie der CANTOS-Studie haben gezeigt, dass Personen mit CHIP, insbesondere solchen mit TET2-Mutationen, überproportional von IL-1β-hemmenden Therapien profitieren [6]. Neben IL-1β-Inhibitoren haben auch andere antiinflammatorische Therapien wie Colchicin positive Ergebnisse gezeigt [13]. Die Integration von CHIP in bestehende Risikomodelle für kardiovaskuläre Erkrankungen könnte dazu beitragen, dass gefährdete Personen frühzeitiger identifiziert und gezielte Präventionsmaßnahmen eingeleitet werden können.
Translationale Perspektiven für Forschung und Klinik
Kondensiert stellt CHIP einen neuen, klinisch relevanten und häufig auftretenden kardiovaskulären Risikofaktor dar. In den aktuellen Leitlinien gibt es bislang keine Empfehlung für ein gezieltes Screening oder eine spezifische Therapie. Häufige Mutationen in Genen wie TET2, DNMT3A und ASXL1 tragen wesentlich zur Entwicklung von Entzündungsprozessen bei, die eine Schlüsselrolle in der Pathogenese von Atherosklerose und anderen kardiovaskulären Erkrankungen spielen.
Fortschritte in der Sequenzierungstechnologie, insbesondere durch NGS mit hoher Sequenziertiefe, könnten zukünftig nicht nur die Identifikation kleinerer klonaler Populationen ermöglichen, sondern auch kosteneffizient gestaltet werden, was eine breitere Anwendung in klinischen Routinen fördert. Dies könnte dazu beitragen, Betroffene frühzeitig zu identifizieren und präventive Maßnahmen einzuleiten. Insbesondere bei Personen mit zusätzlichen Risikofaktoren wie Hypertonie, Diabetes mellitus oder Dyslipidämie könnte eine Integration von CHIP in bestehende Screening- und Risikomodelle das Management kardiovaskulärer Erkrankungen optimieren.
Die zukünftige Forschung sollte nicht nur auf die Charakterisierung häufiger Mutationen abzielen, sondern auch seltene genetische Veränderungen und deren pathophysiologische Relevanz untersuchen. Zudem sollte sie die Entwicklung zielgerichteter Therapien vorantreiben. Die Hemmung spezifischer inflammatorischer Signalwege wie des NLRP3-Inflammasoms oder die Modulation der klonalen Expansion sind dabei erste vielversprechende Ansätze.
Die perspektivische Integration von CHIP in klinische Entscheidungsprozesse hat das Potenzial, die Morbidität und Mortalität von kardiovaskulären Erkrankungen signifikant zu senken. Durch die Kombination aus genetischem Screening, personalisierter Therapie und einem konsequenten Risikofaktorenmanagement können nachhaltige Verbesserungen in der Lebensqualität von Betroffenen erreicht werden. CHIP eröffnet somit nicht nur neue Perspektiven in der Prävention und Behandlung von CVD, sondern stellt auch eine Chance dar, personalisierte Medizin zu fördern.
Interessenkonflikt:
PD Dr. Dr. med. Moritz von Scheidt ist Vorstand des gemeinnützig arbeitenden Vereins Deutsches CHIP Register (www.chip-register.de), das aktuell über 3.500 Individuen mit CHIP-Mutationen unentgeltlich informiert und longitudinal betreut.