Muster in der Datenflut
In-vitro-Diagnostik
Ob Laboratoriumsmedizin, Mikrobiologie oder Pathologie: Viele diagnostische Disziplinen treibt die Sorge um, in den Massendaten zu ertrinken, die zum Beispiel beim Einsatz von Hochdurchsatzsystemen in der Routinediagnostik, bei der Analyse bakterieller Mikrobiome (S. 18) oder bei der Auswahl geeigneter Proben aus Millionen von Biobankproben (S. 12) anfallen.
Wer hier mit Zahlenkolonnen hantiert, hat meist schon verloren. Deshalb nimmt in letzter Zeit die Zahl der Publikationen zu, die ihre Ergebnisse in Form von bunten Mustern präsentieren. Ein Bild sagt nämlich nicht nur mehr als tausend Worte, sondern auch mehr als tausend Zahlen. Mithilfe der Bioinformatik kann man heute medizinische Massendaten so aufbereiten und visualisieren, dass das Auge auf das Wesentliche gelenkt wird.
Das Bild im Hintergrund zeigt das an einem anschaulichen Beispiel: Ein See besteht aus unzähligen, scheinbar ungeordneten Wassertropfen, aber am Muster der Lichtreflexe kann man gut erkennen, ob die Oberfläche von einer ruhigen Brise bewegt oder durch Sturm aufgewühlt wird. Ganz ähnlich bestehen genomische, proteomische oder mikrobiomische Datensätze aus unüberschaubar vielen Einzelwerten, die man per Computer in farbige Muster umwandeln muss, um beispielsweise auf einen Blick diejenigen Biomarker zu identifizieren, die zwischen Gesundheit und Krankheit unterscheiden könnten.
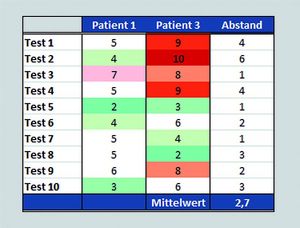
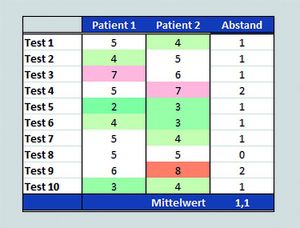
Die Umwandlung von Laborwerten in solche Muster folgt einem mittlerweile gut standardisierten Ablauf, der hier vereinfacht beschrieben werden soll. Zunächst normiert man alle Originaldaten auf eine einheitliche Skala und hinterlegt sie farbig (Abb. 1), um sie unabhängig vom Testverfahren rechnerisch und optisch vergleichbar zu machen. Allein durch diese simple Vorverarbeitung, die im Prinzip jedes Labor-EDV-System automatisieren kann, erkennt man sofort, dass Patient 1 weniger krank zu sein scheint als Patient 3, und dass sich die Werte von Patient 1 und 2 stärker ähneln als die von Patient 1 und 3.
Im zweiten Schritt geht es darum, ein Maß für diese Ähnlichkeit zu finden, um damit rechnen zu können. Am einfachsten und effektivsten ist es, die absoluten Differenzen zwischen jeweils zwei benachbarten Werten zu bilden (rechte Spalte). Der Mittelwert dieser Abstände ist im oberen Bild 1,1 und im unteren 2,7. Also ähneln sich Patient 1 und 2 viel stärker als Patient 1 und 3, was den optischen Eindruck bestätigt.
Im dritten und letzten Schritt, dessen Ergebnis im Hintergrundbild zu sehen ist, lässt man den Computer alle Fälle aufgrund dieser Ähnlichkeit sortieren, sodass sich gleiche Farben in sog. Hotspots anhäufen (Clustering). Der eingerahmte hellgrüne Bereich signalisiert zum Beispiel, dass die linken acht Fälle (Patienten, Bakterien oder was auch immer) eine auffällige Ähnlichkeit hinsichtlich der obersten sechs Biomarker aufweisen.
Die Bewertung solcher Befunde bleibt natürlich Sache des Arztes bzw. Wissenschaftlers; aber ohne die beschriebenen Algorithmen wäre eine Interpretation von Massendaten völlig unmöglich. Es ist deshalb zu erwarten, dass die Mustererkennung neben der reinen Betrachtung von Zahlen immer größere Bedeutung erlangen wird.
gh